

论文标题:Attenuation of Piwil2 induced by hypoxic postconditioning prevents cerebral ischemic injury by inhibiting CREB2 promoter methylation
发表期刊:Brain Pathology(IF=7.611)
研究方法:RIP-seq、piRNA 测序、qPCR 等
1、Piwil2 下调起到了神经保护作用
作者准备了4组实验大鼠,其中两组未进行手术处理,剩余两组都经过了短暂全局性脑缺血(tGCI)的手术处理。在未进行手术处理的大鼠当中,其中一组大鼠经历了低氧条件(Hypoxia),另一组则没有经历低氧条件(作为对照组)。在进行了 tGCI 手术处理的大鼠当中,其中一组大鼠在其后经历了低氧后处理(HPC),而另一组大鼠则没有经历 HPC 低氧后处理。作者随后将这些大鼠大脑海马当中的 CA1 区取出,针对 Piwil2 蛋白进行了免疫组化染色以及 Western Blot 检测,发现 tGCI 处理可以增加 Piwil2 蛋白的表达,而 HPC 处理则可以减弱 tGCI 对 Piwil2 蛋白表达的增强作用;针对 Piwil2 mRNA 的 RT-qPCR 也呈现出类似的结果。作者通过设计荧光融合蛋白,认定 Piwil2 的亚细胞定位主要在神经元细胞核(NeuN)中;在 tGCI 组中,Piwil2 蛋白主要位于星形胶质细胞当中;而在 HPC 组中,Piwil2 蛋白主要位于神经元细胞当中。
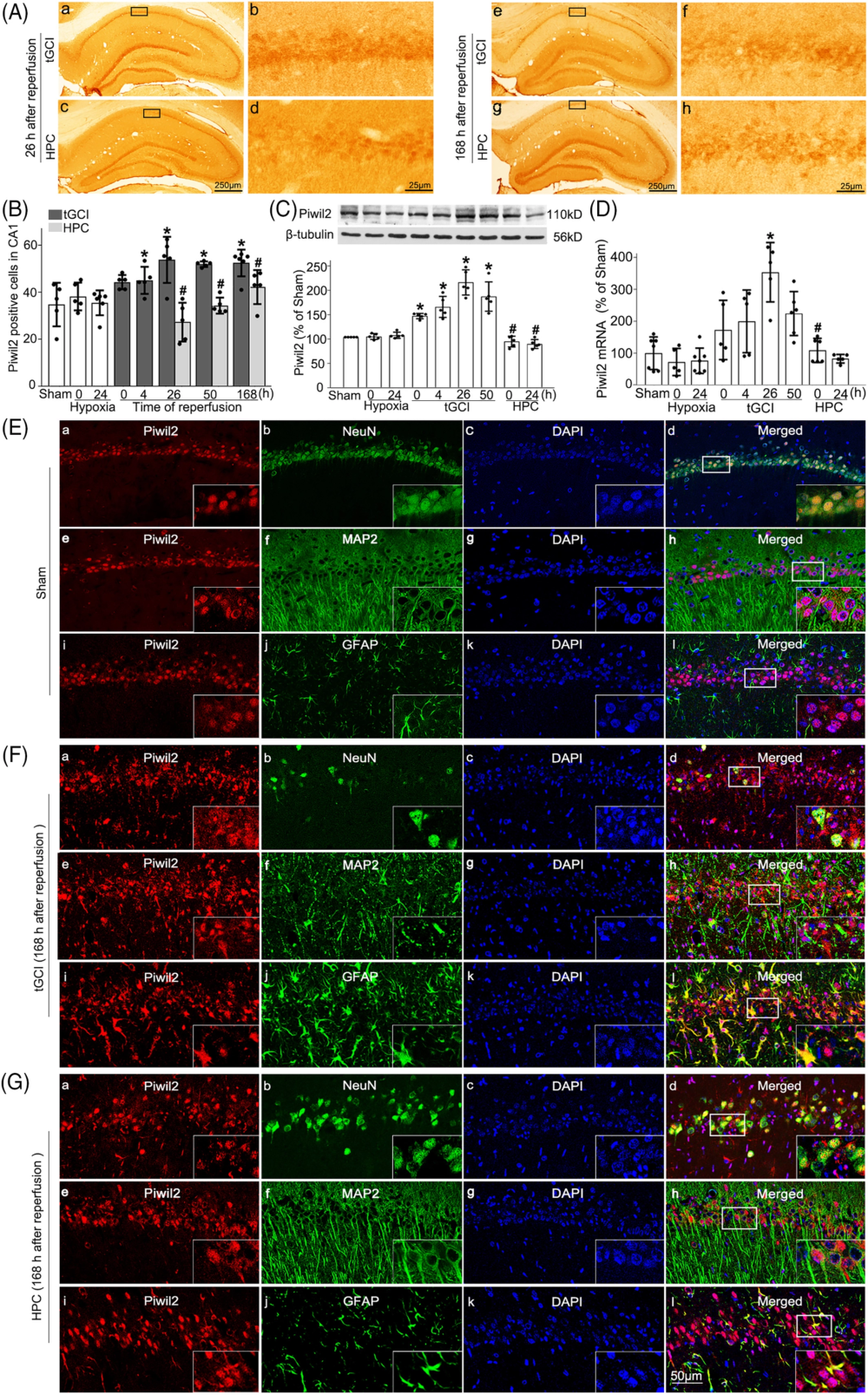
综上所述,HPC 后在 CA1 区的 Piwil2 蛋白下调起到了拮抗 tGCI 效应的神经保护作用。
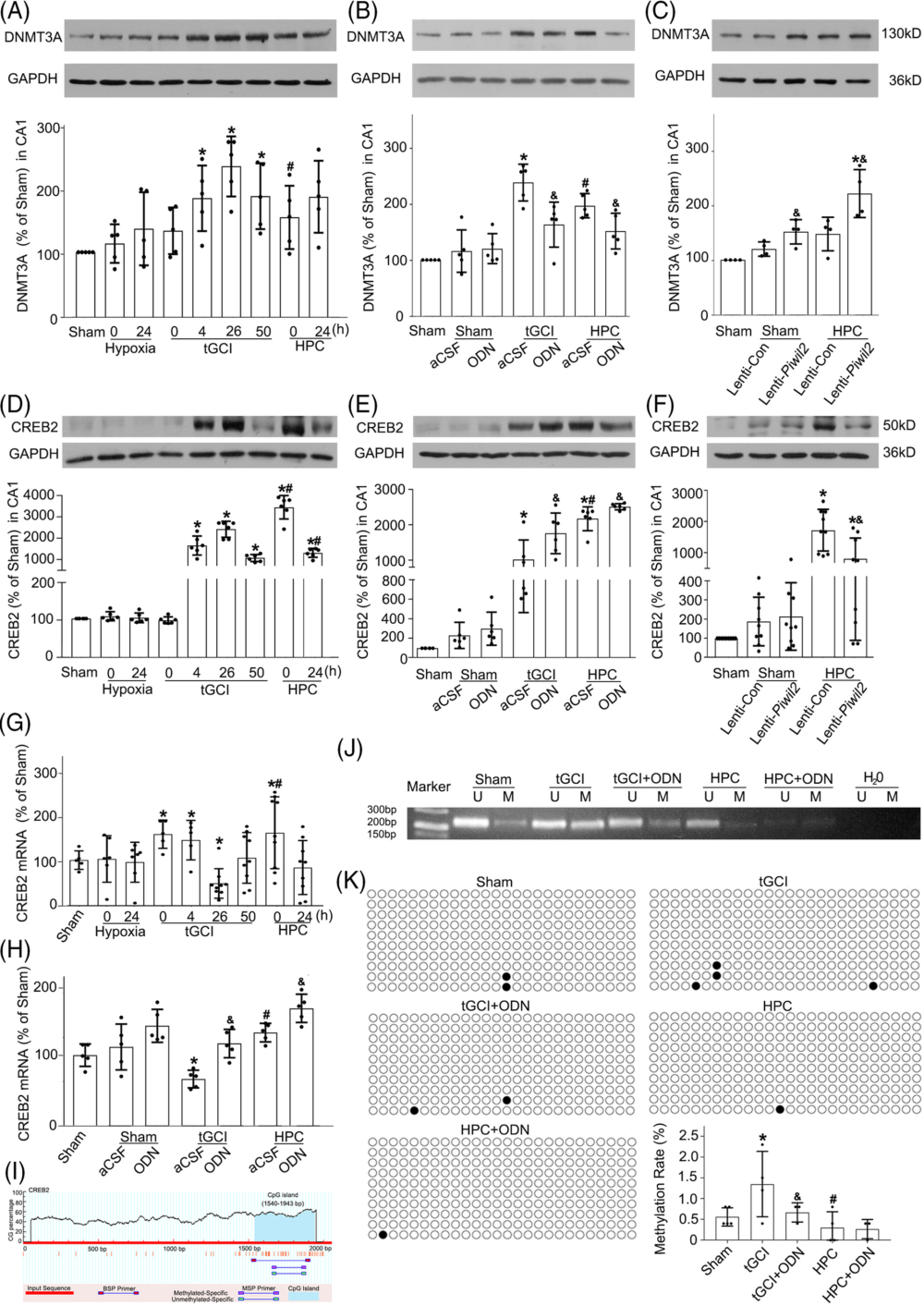
2、Piwil2/piRNAs 通路靶向促进 DNMT3A 表达
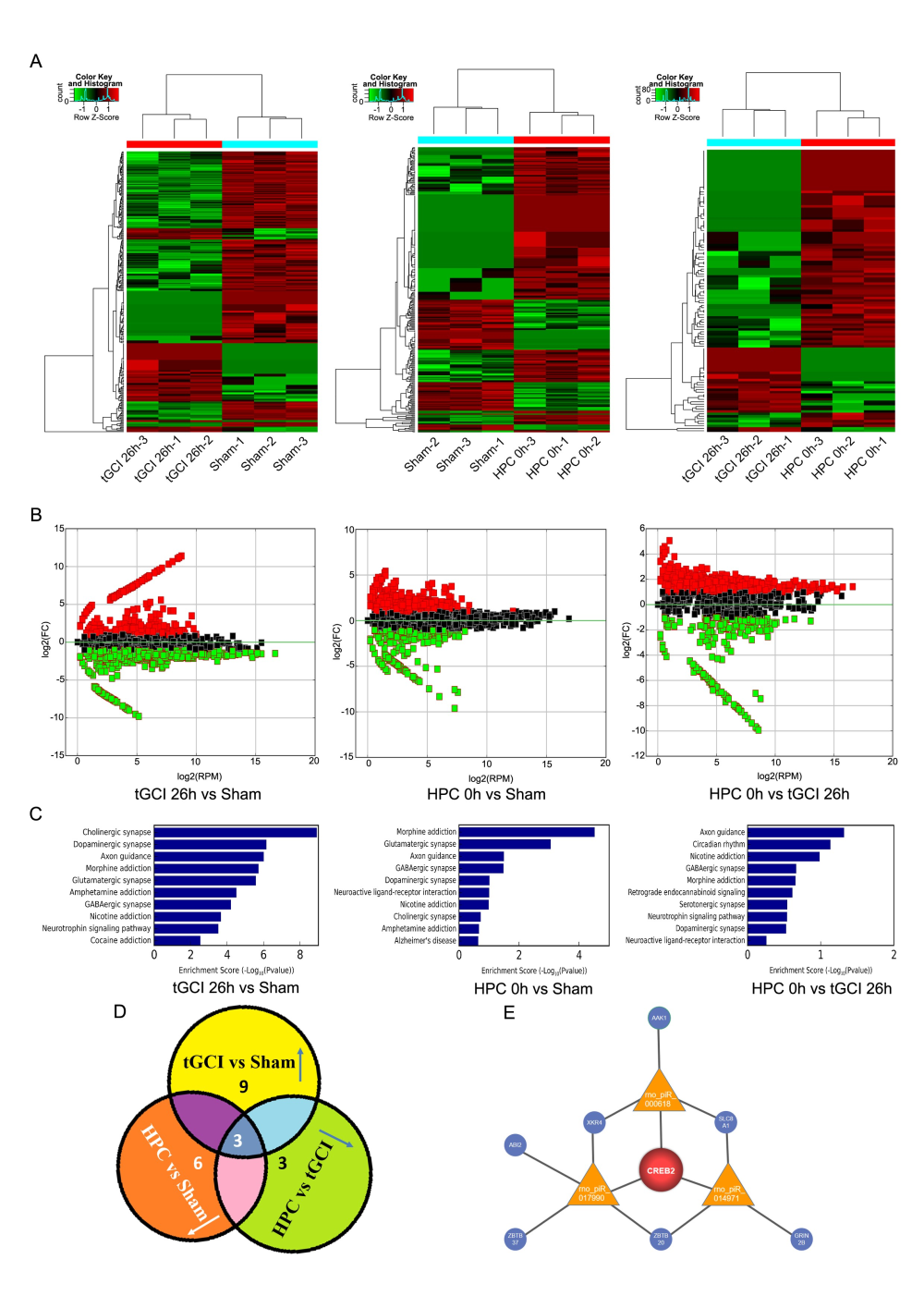
作者对 tGCI 处理组、tGCI+ HPC 处理组以及对照组的大鼠脑 CA1 区进行了 piRNA 测序,发现各组之间的 piRNA 谱存在系统性的差异。组间差异 piRNA 主要富集于轴突引导(axon guidance)和突触功能(synaptic functions)相关的 KEGG 通路当中。有 12 个 piRNA 在 tGCI 组当中相比于对照组存在显著差异上调,有 6 个 piRNA 在 HPC 组中相比于 tGCI 组存在显著差异下调,有 9 个 piRNA 在 HPC 组中相比于对照组存在显著差异下调——在这三部分 piRNA 当中,有 3 个 piRNA 重复出现,它们是:rno_piR_000618、 rno_piR_017990 和 rno_piR_014971。生物信息学分析还显示,CREB2 是这三个 piRNA 共有的靶基因。

3、Piwil2 通过促进 DNA 甲基化来抑制 CREB2 的表达
至于先前发现的三个 piRNA 的共有靶基因 CREB2,它的蛋白水平在 tGCI 后呈现先上升、后下降的现象;而 HPC 处理则可以维持 CREB2 蛋白的高水平。CREB2 在 mRNA 水平也表现出相似的现象。由于 Piwi/piRNA 已知可以参与 DNA 甲基化,作者针对 CREB2 基因的 DNA 预测出了其启动子上 CpG 岛的存在,并通过 MSP 和 BSP 两种甲基化 PCR 方法验证了这些 DNA 甲基化位点的真实存在。其中,tGCI 组的 DNA 甲基化程度最高,对照组其次,而 HPC 处理后 DNA 甲基化程度最低。为了验证 Piwil2 蛋白对于 CREB2 基因甲基化的必要性,作者还进行了 Piwil2 敲降,结果发现 tGCI 导致的 DNA 甲基化程度升高现象被逆转。综上所述,作者认为HPC 诱导的 Piwil2 下调通过抑制 DNA 甲基化来增强 CREB2 的。
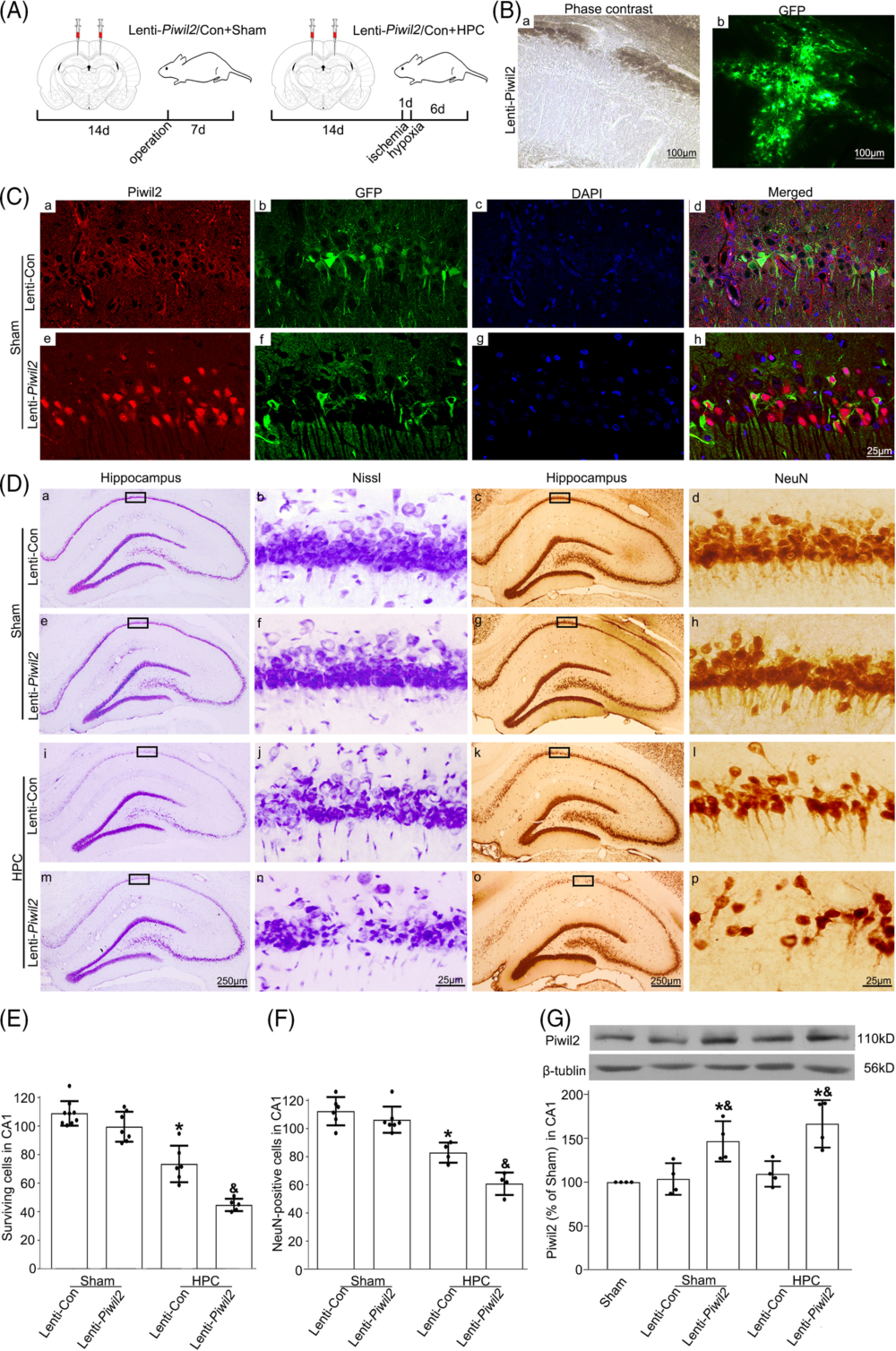
4、Piwil2 下调可改善神经元可塑性以及记忆功能
作者在 tGCI 处理后一段时间对大鼠脑的海马区域进行了高尔基染色(Golgi staining),发现 CA1 区的椎体神经元遭到了严重损坏,但是如果在 tGCI 后进行了 HPC 处理或 Piwil2 敲降,则损坏的程度减轻。Sholl analysis 结果也显示,HPC 处理或 Piwil2 敲降组当中的树突分支交叉数目高于 tGCI 组,这一差异在距离神经元细胞体 80 到 200 μm 处的树突分支交叉体现得尤为明显。细胞形态分析也显示,tGCI 组的树突长度以及树突棘密度都比对照组下降,但 HPC 处理或 Piwil2 敲降则能减轻这一现象。为了检验神经元的这些变化是否影响大鼠的记忆和认知能力,作者还对大鼠进行了 Morris 水迷宫测试(Morris Water Maze),发现 tGCI 组大鼠的逃脱路径长度(path length)以及逃脱用时(excape latency)都长于对照组,而 HPC 组大鼠和 Piwil2 敲降组的大鼠则在经过数日训练后降低了逃脱路径长度和逃脱用时,而不同组别的大鼠在游泳速度上并无差异,说明逃脱能力上的差异是由于认知能力差异而非体力差异造成的。综上所述,HPC 诱导的 Piwil2 下调增强了海马区神经元树突棘的可塑性并起到改善记忆功能的作用。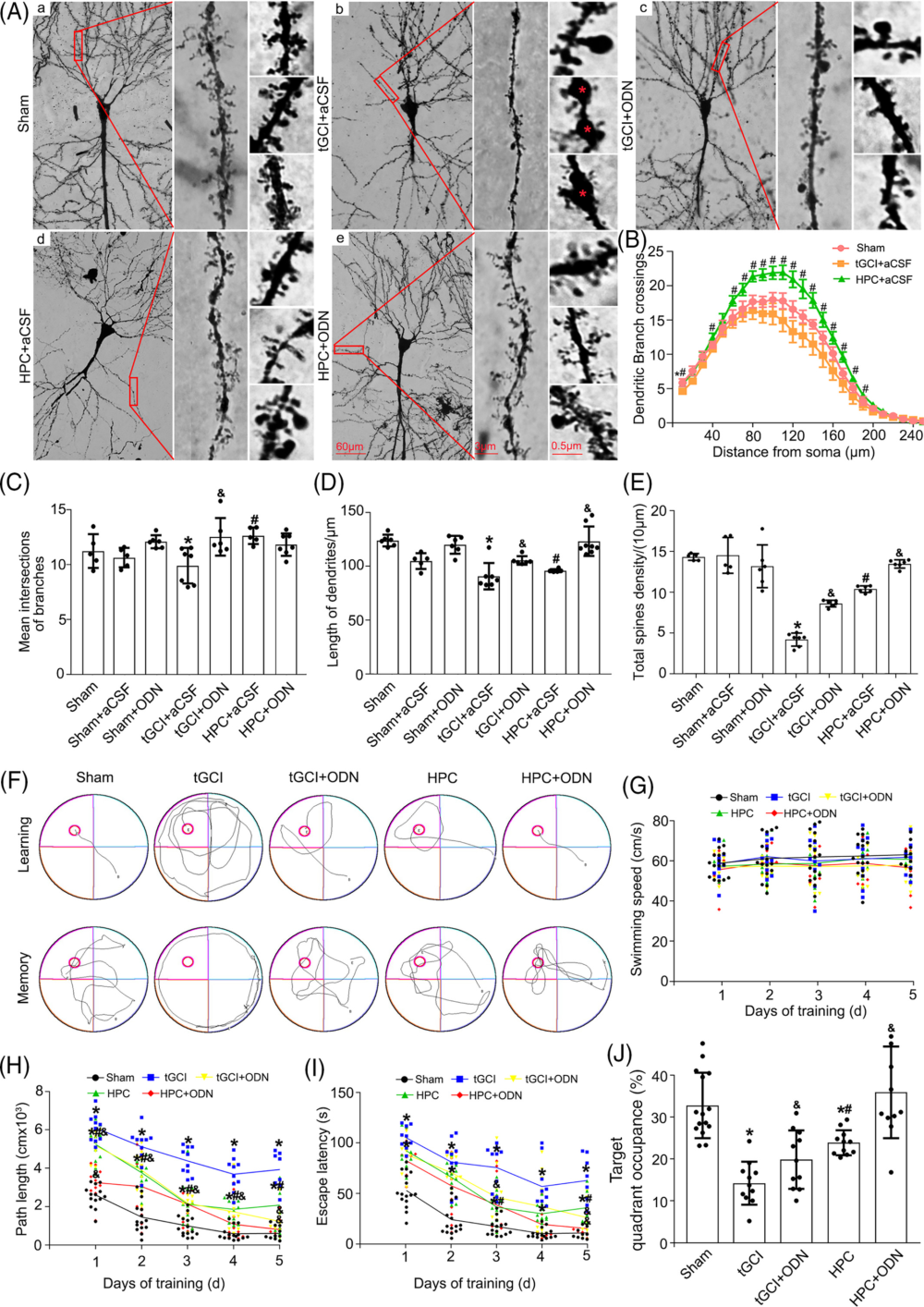
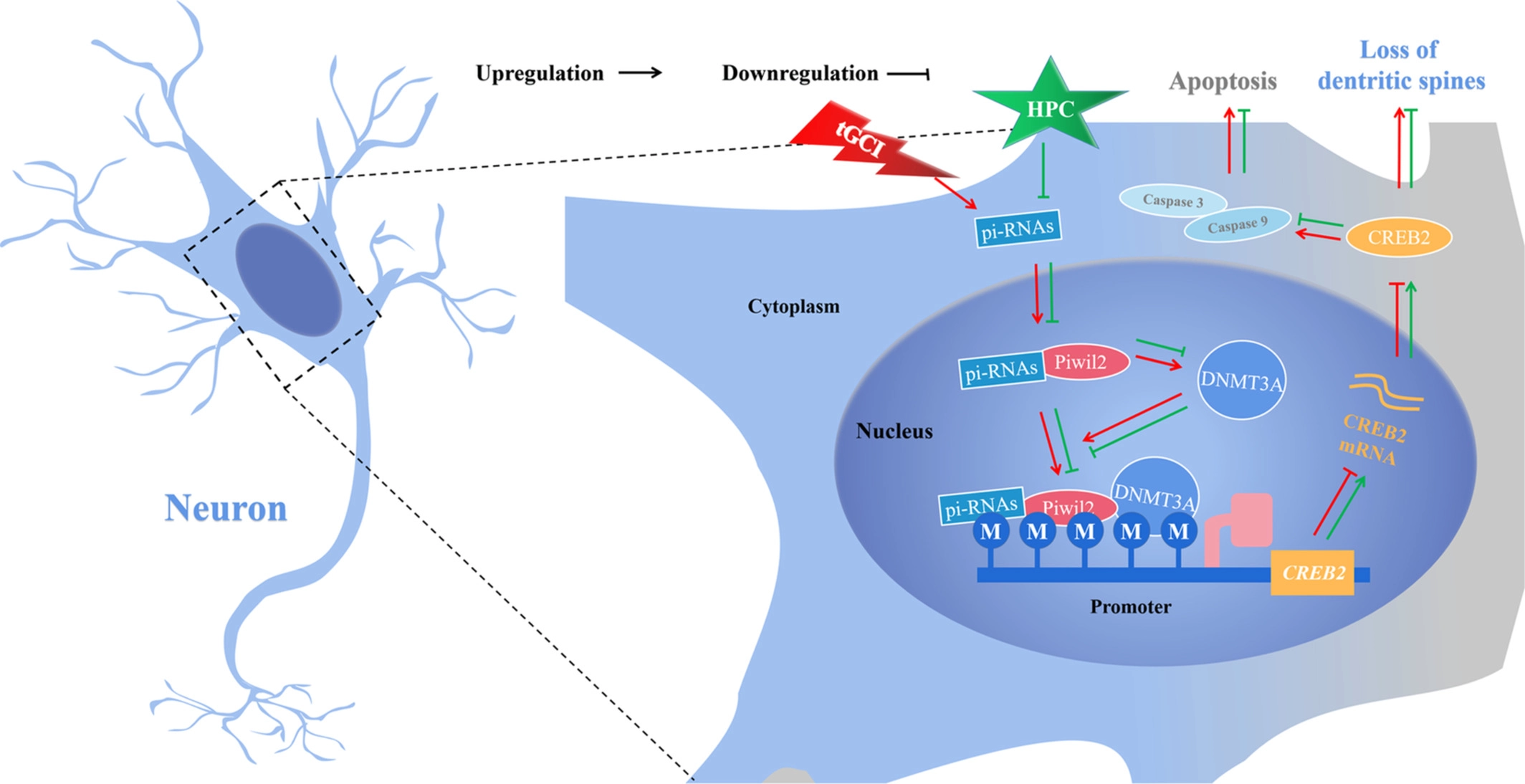
在本研究中,作者描述了 Piwil2在通过表观遗传机制调节 CREB2表达和介导大鼠 tGCI 后 HPC 的神经保护中的作用。HPC 下调了 tGCI 后 CA1区域中 Piwil2的表达,并降低了 DNA 甲基转移酶 DNMT3A 的水平,DNMT3A 反过来消除了 tGCI 诱导的 CREB2启动子甲基化的增加,从而增加了 CREB2在 mRNA 和蛋白质水平。此外,HPC 诱导的 Piwil2的减少起到了恢复树突复杂性和树突长度的作用,并阻止了 CA1区域神经元树突棘的丧失,从而改善了 tGCI 后大鼠的学习和记忆功能。
优势一:云序累计支持客户发表 240+ 篇SCI论文,合计影响因子近 2100 分,其中包含多篇高分小 RNA 测序类论文。
优势二:全面检测各类非编码RNA(piRNA, miRNA, tRNA, tiRNA&tRF, 细菌sRNA,circRNA,lncRNA,Pri-miRNA等)。
优势三:能够一站式提供非编码小RNA的机制研究,RIP测序,RNA pulldown,ChIP测序等多种服务,助力用户解决机制研究
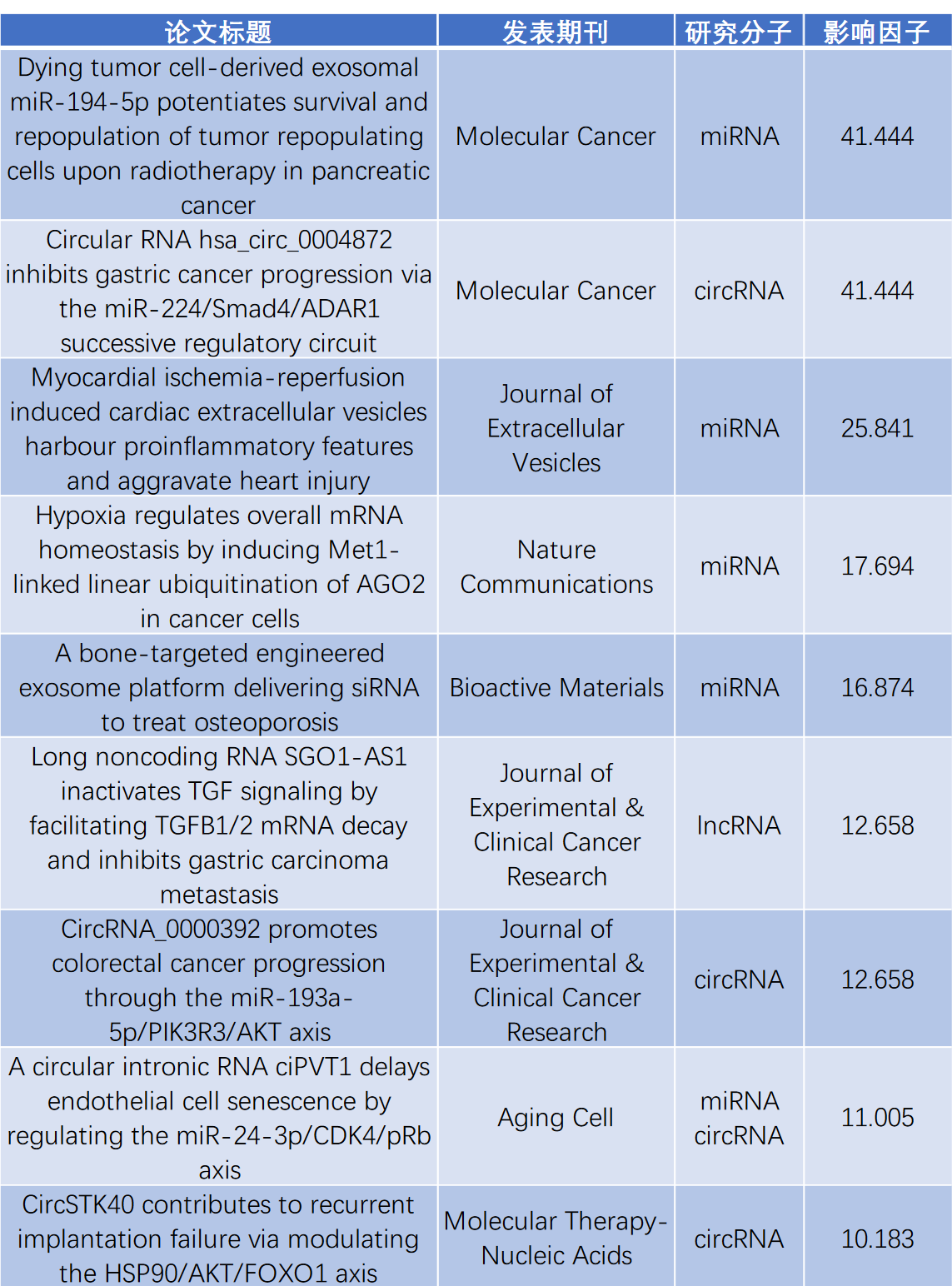
云序客户非编码 RNA 部分高分论文
相关产品
往期回顾

