说到水稻,大家肯定会想到1月份上海师范大学生命与环境科学学院黄学辉教授团队发表在《Nature Genetics》上的文章[1]。
水稻是世界上重要的粮食作物,其基因组序列信息也是作物中最早公布的。目前水稻中经典的参考基因组有粳稻日本晴和籼稻93-11。虽然这两种基因组数据在不断完善和升级,但仍无法完全覆盖水稻丰富多样的遗传信息。要知道有的植物品种间的遗传差异甚至比人和猩猩的差异都大……
研究人员选取了66份来自不同水稻类群的栽培稻和野生稻材料作为研究对象,对这些水稻品系进行深度测序和de novo组装后,获得其精细基因组图谱及遗传变异并深入分析。
研究不仅完善了栽培稻和野生稻的泛基因组数据集信息,还发现了可能具有生物功能的新基因及许多新SNP位点和InDels(插入缺失突变)。研究成果将对功能基因组的深入研究以及分子育种有重要作用。
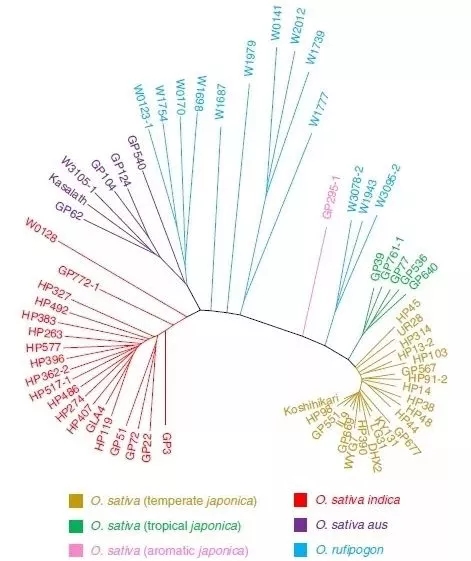
66个水稻品种的系统发育树图
“一个物种基因组计划的完成,就意味着这一物种学科和产业发展的新开端。”水稻基因组的完善,也意味着水稻有更多的研究方向等待我们去发掘。

研究方法:重测序——变异检测
2017年10月,华中农业大学邢永忠教授团队在《Nature Plants》发表了关于水稻的文章[2]。研究发现水稻的穗发育基因(FRIZZY PANICLE,FZP)在自然界中发生变异,可显著增加水稻每穗的粒数和轻微降低每粒的粒重,使水稻产量增加15%。
邢永忠研究小组就利用粒形差异显著的亲本的 FZP上游5.3kb处,发现一个18bp片段的转录沉默子发生复制,造成拷贝数变异 (Copy number variation,CNV),名为CNV-18bp,使得有两个18bp片段串联在一起。研究发现有两个18bp片段的水稻千粒重大概减少了10%左右,每穗粒数增加 40%-50%,穗数并无显著性差异,但水稻产量要比只有一个18bp片段的高15%。
事实上,绝大多数水稻中只有一个18bp片段,只有印度、孟加拉等东南亚地区的少数品种才有两个串联的18bp片段。这种是在自然界中发生变异的。因此,可以利用分子标记辅助育种,使该优良等位基因在中国高产品种的改良中发挥巨大作用。

华中农大——邢永忠教授
研究方法:重测序—群体进化
2017年5月,浙大樊龙江教授团队在《Nature Communications》上发表了文章[3]。去驯化是栽培作物从人工环境返回自然环境的遗传现象,目前大多都关注于作物的驯化,然而对去驯化的研究却很少。杂草稻(Oryza sativa f.spontanea)是研究去驯化分子过程的经典模型。研究通过对155份杂草稻和76份当地栽培稻进行全基因组测序(平均深度18X),研究杂草稻去驯化的环境适应性机制。
系统发育和群体遗传分析表明,我国杂草稻均起源于栽培稻,在起源过程中均经历了强烈的遗传瓶颈效应。基因组分化分析分别鉴定出了四个主要杂草稻群体和去驯化相关的位点,并且在7号染色体6.0~6.4Mb区间发现了各独立起源杂草稻的趋同进化区域。杂草稻基因组上发现很多区域受到了平衡选择信号,这表明平衡选择后产生的遗传多态性可以适应复杂的生存环境。
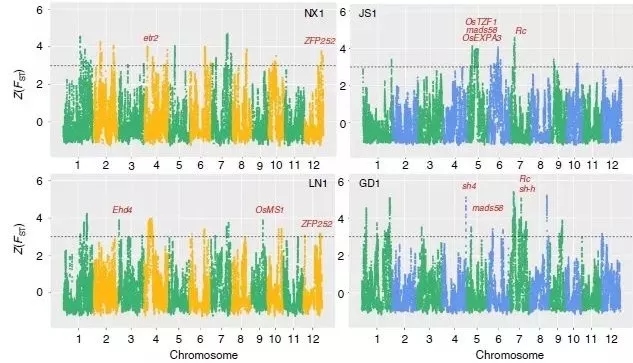
来自宁夏(NX1)和辽宁(LN1)的粳稻杂草稻以及来自江苏(JS1)和广东(GD1)的籼稻杂草稻的功能基因差异
研究方法:重测序—GWAS
2018年1月,《Frontiers in Plant Science》在线发表了中国水稻研究所魏兴华研究员研究团队关于水稻全基因组关联分析(GWAS)的文章[4]。
种子休眠是影响作物产量品质的重要农艺性状,主要受环境条件影响和遗传因素控制。研究利用全基因组关联的方法对水稻休眠相关性位点进行了深度挖掘。通过关联分析,在水稻的其中8条染色体上共找到了9个显著性SNP位点,可以关联34.9%的表型变异。通过候选基因预测,共找到了212个休眠性候选基因。进一步分析表明,9个中有3个显著性位点位于这些驯化区域附近。该研究通过对种子休眠性位点的深入挖掘和驯化位点的深入分析,对将来解析种子休眠性分子机制和研究水稻基因组人工驯化具有重要意义。
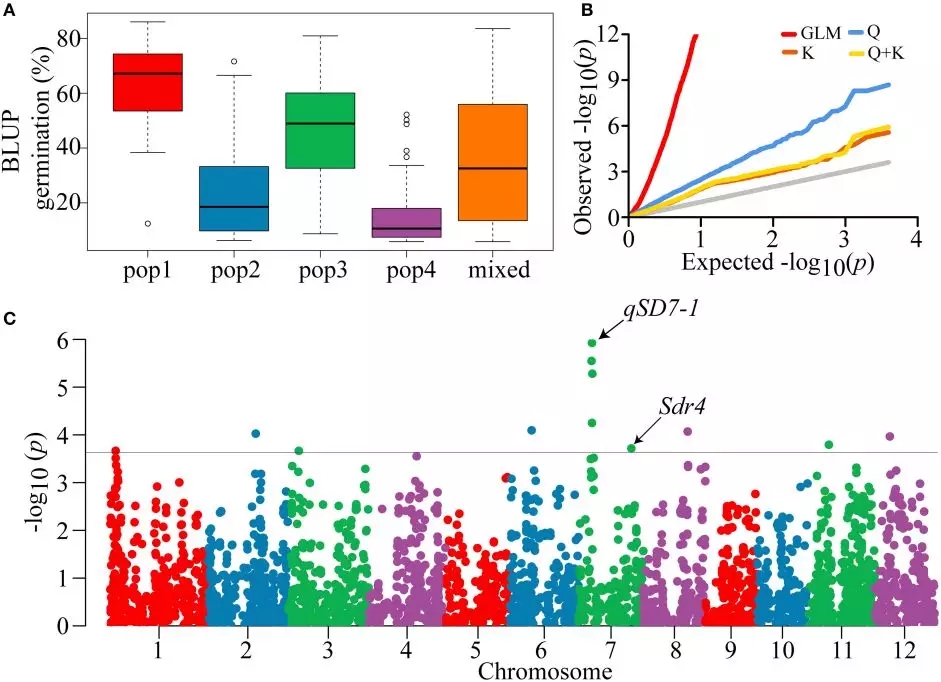
种子休眠的表型分布和GWAS
物种基因组信息的完善,有助于我们通过重测序的方法去发掘更多藏在其他品种基因组里的“惊喜”。博奥晶典不仅可以提供基因组De novo的服务,也提供完整的动植物重测序方案,研究手段更是涵盖了变异检测、BSA性状定位、遗传图谱构建、GWAS、群体进化。对于复杂的结构变异和拷贝数变异,更有10X Genomics的linked-read测序方法来解决困难!
参考文献
[1] Qiang Zhao, Qi Feng,et al. Pan-genome analysis highlights the extent of genomic variation in cultivated and wild rice. Nature Genetics,2018.
[2] Xufeng Bai, Yong Huang,et al. Duplication of an upstream silencer of FZPincreases grain yield in rice. Nature Plants,2017.
[3] Qiu J, Zhou Y, Mao L, et al. Genomic variation associated with local adaptation of weedy rice during de-domestication.Nature Communications, 2017.
[4] Qing Lu,Xiaojun Niu, et al. Genome-Wide Association Study of Seed Dormancy and the Genomic Consequences of Improvement Footprints in Rice (Oryza sativa L.). Frontiers in Plant Science,2018.
博奥晶典科研服务事业部 李楠、李超、李媛媛 | 文案
部分配图来源于网络 侵删
